|
Hallo, ich versuche aktuell die Strukturformel von Xenontetrafluorid mit Chemfig in mein Dokument einzubinden. Öffne in Overleaf
\documentclass{standalone} \usepackage{chemfig} \begin{document} \chemfig{F-Xe(-[:90]F)(-[:-90]F)-F} \chemfig{F*4(-F-F-F-)} \end{document} Das sind die beiden Ansätze die ich bisher habe. Ich würde nun gerne die Fluoratome miteinander verbinden, um die quadratische Form hervorzuheben. Bei meinem ersten Versuch weiß ich nicht wie ich die Bindungen zwischen den Fluoratomen reinbekomme, weshalb ich es beim zweiten Versuch mit einem Ring versucht habe. Da bekomme ich das Xenon aber nicht in die Mitte gebracht und verbunden. (Wie) Lässt sich das irgendwie anders umsetzen? |
|
Chemfig erlaubt es einem, relativ leicht den Nodes Namen zu geben mit der Öffne in Overleaf
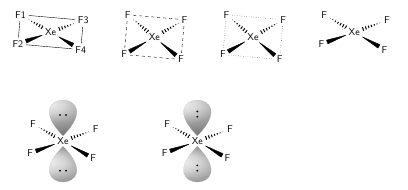
\documentclass[border=3.14mm]{standalone} \usepackage{chemfig} \begin{document} \chemfig{@{F1}F-Xe(-[:90]@{F2}F)(-[:-90]@{F3}F)-@{F4}F} \tikz[overlay,remember picture]{% \draw[densely dotted](F1)--(F2)--(F4)--(F3)--(F1);} \chemfig{@{f1}F*4(-@{f2}F-@{f3}F-@{f4}F-)} \tikz[overlay,remember picture]{% \node at (barycentric cs:f1=1,f2=1,f3=1,f4=1) (Xe) {Xe}; \foreach \X in {1,...,4} {\draw(f\X)--(Xe);} } \end{document} Ich habe im ersten Molekül die Linien hier gepunktelt gezeichnet, um sie von Bindungen zu unterscheiden, aber Du kannst |
|
So wie ich das Probelm verstehe, soll wohl die Ebene der Flouratome dargestellt werden. Dazu muss man die perspektivisch richtigen Bindungen wählen und die Winkel dazwischen. Zeichnet man die noch die beiden am Xenon vorhandenen Elektronenpaare ein, dann entsteht ein halbwegs perspektivisch korrektes Molekül, ohne dass man die quadratisch planare Form mit Linien hervorheben muss. \documentclass{article} \usepackage{chemfig,chemmacros} \chemsetup{modules=all} \renewcommand*\printatom[1]{\ensuremath{\mathsf{#1}}} \setchemfig{atom sep = 3.5em, cram width = 4pt, bond offset = 3pt, } \chemsetup[orbital]{ overlay, opacity = .75, p/scale = 2.4, p/color = gray!60, } \begin{document} \chemfig{F1?[a]?[d]<:[:-30]Xe(<[:-160]F2?[a]?[c])(>:[:20]F3?[b]?[d])<[:-30]F4?[b]?[c]} \hspace{1cm} \chemfig{F?[a]?[d]<:[:-40]Xe(<[:-150]F?[a,,,dashed]?[c])(>:[:30]F?[b]?[d,,,dashed])<[:-40]F?[b,,,dashed]?[c,,,dashed]} \hspace{1cm} \chemfig{F?[a]?[d]<:[:-40]Xe(<[:-150]F?[a,,,dotted]?[c])(>:[:30]F?[b]?[d,,,dotted])<[:-40]F?[b,,,dotted]?[c,,,dotted]} \hspace{1cm} \chemfig{F<:[:-30]Xe(<[:-160]F)(>:[:20]F)<[:-30]F} \par\vspace{1.5cm} \chemfig{F<:[:-30]Xe(-[6,.05,,,draw=none]{\orbital[half,angle=-90]{p}})(-[6,.8,,,draw=none]{\lewis[lewis dist=6pt]{2:,}})(-[2,.25,,,draw=none]{\orbital[half,angle=90]{p}})(-[2,.8,,,draw=none]{\lewis[lewis dist=6pt]{2:,}})(<[:-160]F)(>:[:20]F)<[:-30]F} \hspace{2cm} \chemfig{F<:[:-30]Xe(-[6,.05,,,draw=none]{\orbital[half,angle=-90]{p}})(-[6,.8,,,draw=none]\lewis{2.,})(-[6,.65,,,draw=none]\lewis{2.,})(-[2,.25,,,draw=none]{\orbital[half,angle=90]{p}})(-[2,.9,,,draw=none]\lewis{2.,})(-[2,.75,,,draw=none]\lewis{2.,})(<[:-160]F)(>:[:20]F)<[:-30]F}\par\vspace{1.5cm} \end{document} |

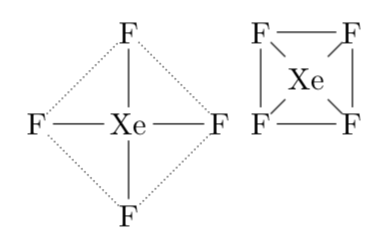
Xenontrifluorid? Deinen Versuchen nach meinst Du Xenontetrafluorid, nicht? Aber welche Bindungen sollen dann fehlen?
Ups, natürlich sollte es "tetra" sein, hab davor mit Bortrifluorid gearbeitet... Mit "fehlenden Bindungen" hab ich mich wohl auch falsch ausgedrückt, chemisch fehlen zwischen den Fluoratomen keine Bindungen, ich würde die aber gerne aus geometrischen Überlegungen zur Symmetrie des Moleküls einzeichnen, um die quadratische Form hervorzuheben.